DNAオリガミから2次元自己集合体を作成:医療技術ニュース
京都大学の研究グループは、DNAオリガミからマイクロメーターサイズの2次元集合体を作成する技術を開発した。多様なパターンの集合体作成が可能で、複数種の生体分子を配置することもできる。
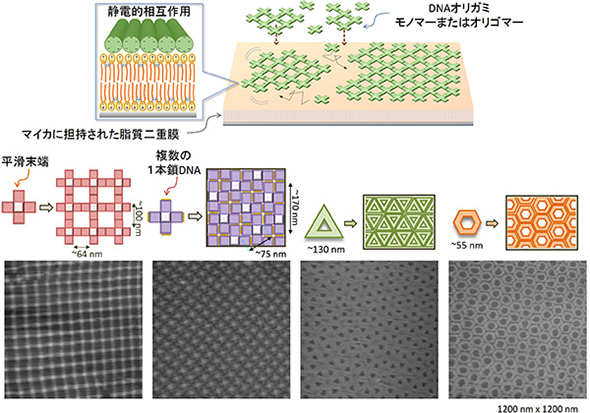
京都大学物質−細胞統合システム拠点(iCeMS)の遠藤政幸准教授、同大大学院理学研究科の鈴木勇輝特定研究員、iCeMS・理学研究科の杉山弘教授の研究グループは2015年8月28日、DNAオリガミと呼ばれる約100nmのDNA平面構造体からマイクロメーターサイズの2次元自己集合体を作成する技術を開発したと発表した。
研究グループが開発したのは、DNAオリガミを人工脂質二重膜表面に吸着・濃縮し、2次元自己集合化させることで多様な空間パターンのDNA自己集合体を創出する方法だ。マイカと呼ばれる平面の基盤上に脂質二重膜を作成し、そこへ一辺が100nmの十字型DNAオリガミ構造体(モノマー)を静電的に吸着すると、モノマー同士が結合し、格子構造を形成した。
また、高速原子間力顕微鏡(高速AFM)によって、DNAオリガミの単量体や多量体が膜上で結合・解離をくり返しながら大型の格子構造へ成長していく様子を実時間でとらえた。加えて、格子中の点欠陥が溶液中の単量体によって補填・修復される様子も観察できた。
さらには、DNAオリガミの形状やオリガミ間の相互作用の様式を変更することで、多種多様なパターンの集合体が作成できることを確認。集合体表面の任意の部分にさまざまな外来分子を装飾・配置することにも成功した。
今回の研究の成果は、ボトムアップ型ナノテクノロジーの基盤技術となるもので、ナノデバイスの集積化、組織化や新規分子デバイス構築などへの展開・応用が期待される。
- 減らすだけでは効果なし、完全禁酒/禁煙で食道がん治療後の発生リスクが5分の1に
- 既存薬が効きにくいがんにも光、BNCT用ホウ素薬剤を開発
- 不公平な提案を受諾する際の脳内抑制メカニズムを解明
- 新型コロナ向け「ワクチン接種パッチ」の実用化へ前進、自己接種も可能に
- なぜPM2.5で花粉症がひどくなる? スズと粘液の反応によるメカニズムを発見
- 先天性の「足の臭い」、長島型掌蹠角化症の原因菌と有効治療法を発表
- ナノイー技術で花粉症状を緩和、パナソニックがヒト臨床試験で実証
- 赤色レーザーダイオードで光合成速度が最大2割向上、植物工場での活用へ
- 健康な社会づくりを目指すミラノ・コルティナ2026とイタリアのAI/サイバー政策
- 卵の段階でひよこの性別を選別する画像認識AIを開発
関連記事
 DNAから染色体を作るための重要な過程を発見
DNAから染色体を作るための重要な過程を発見
東京大学は、ひも状のDNA分子が折り畳まれて染色体が形成される際に重要な役割をもつ反応を発見し、遺伝情報の源であるDNAが細胞の中で安全に保管される仕組みの一端を明らかにしたと発表した。 がん化の原因となるDNAの突然変異の仕組みを解明
がん化の原因となるDNAの突然変異の仕組みを解明
京都大学の武田俊一教授らの研究グループは、DNAの突然変異が引き起こされる仕組みを解明した。従来の説を覆し、複製ポリメラーゼによるコピーでも、突然変異につながるという事実を明らかにした。 ゲノム、遺伝子、DNA、染色体は全て違う
ゲノム、遺伝子、DNA、染色体は全て違う
つまり、専門用語が難しいということ。 食中毒原因菌14種を2時間以内に同時判定するDNA検査装置と検査キット
食中毒原因菌14種を2時間以内に同時判定するDNA検査装置と検査キット
東芝は、新型のDNA検査装置「GenelyzerII」と、14種の食中毒原因菌を同時判定できる「衛生管理用検査キット」を発表した。従来の培養法で4〜5日程度必要だった食中毒原因菌の検出が、2時間以内に同時判定できる。 人工知能「Watson」でがん患者を救う、ゲノム治療の促進へ
人工知能「Watson」でがん患者を救う、ゲノム治療の促進へ
IBMの人工知能「Watson」を、がん治療に役立てるための取り組みが始まっている。Watsonを使って、医学研究成果や医学論文、治療の実績などのデータベースを参照、解析し、患者のDNAに合わせたゲノム治療を、効率的に提案することを目指す。
関連リンク
Copyright © ITmedia, Inc. All Rights Reserved.
医療機器の記事ランキング
- おむつかぶれや尿路感染を防ぐ、体外式カテーテルに新製品
- 3DとAIで数十万の細胞を解析する「自律型デジタル細胞診」システムを開発
- 既存薬が効きにくいがんにも光、BNCT用ホウ素薬剤を開発
- 減らすだけでは効果なし、完全禁酒/禁煙で食道がん治療後の発生リスクが5分の1に
- 「入れ歯」専用の次世代3Dプリンタ、2027年市場投入へ
- 不公平な提案を受諾する際の脳内抑制メカニズムを解明
- 対象所見を10種類に拡充した胸部単純X線画像病変検出ソフトの提供を開始
- 体温の日内制御、恒温動物と変温動物で共通の仕組みを発見
- 新型コロナ向け「ワクチン接種パッチ」の実用化へ前進、自己接種も可能に
- なぜPM2.5で花粉症がひどくなる? スズと粘液の反応によるメカニズムを発見
コーナーリンク